Python在Spark上的机器学习之机器学习实战(上)
MLlib 的使用
在上面的章节之中,我们已经讲过了如何利用PySpark进行数据操作和分析了。那么在这篇文章中,我们就真正利用PySpark结合MLlib来建立一个分类模型。
MLlib :即Machine Learning Library,MLlib 是Spark对常用的机器学习算法的实现库,同时包括相关的测试和数据生成器。MLlib 目前支持四种常见的机器学习问题:二元分类,回归,聚类以及协同过滤,同时也包括一个底层的梯度下降优化基础算法。
载入和转化数据
首先,我们在建立一个DataFrame之前,我们先针对性的指定下DataFrame中数据类型,方便我们数据后期的分析与计算。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
import pyspark.sql.types as typ
labels = [
( 'INFANT_ALIVE_AT_REPORT' , typ . StringType ()),
( 'BIRTH_YEAR' , typ . IntegerType ()),
( 'BIRTH_MONTH' , typ . IntegerType ()),
( 'BIRTH_PLACE' , typ . StringType ()),
( 'MOTHER_AGE_YEARS' , typ . IntegerType ()),
( 'MOTHER_RACE_6CODE' , typ . StringType ()),
( 'MOTHER_EDUCATION' , typ . StringType ()),
( 'FATHER_COMBINED_AGE' , typ . IntegerType ()),
( 'FATHER_EDUCATION' , typ . StringType ()),
( 'MONTH_PRECARE_RECODE' , typ . StringType ()),
...
( 'INFANT_BREASTFED' , typ . StringType ())
]
schema = typ . StructType ([
typ . StructField ( e [ 0 ], e [ 1 ], False ) for e in labels
])
下一步,我们通过 .read.csv() 方法来载入数据,这个方法除了能够载入原数据之外还可以载入GZipped压缩后的csv数据。其实header参数设为 True 代表数据文件的第一行是数据的元信息(即为列表的说明字段)。
births = spark.read.csv('births_train.csv.gz' ,
header=True ,
schema=schema)由于在我们的数据集中有大量的分类变量都是字符串,所以我们首先要想办法将这一类变量转换成数字的形式。
# 转换'INFANT_ALIVE_AT_REPORT'
recode_dictionary = {
'YNU' : {
'Y' : 1 ,
'N' : 0 ,
'U' : 0
}
}在这里总的来说,我们的目的就是一个二分类问题,即预测婴儿的存活情况,也就是“存活 1 ”或“死亡 0 ”。因为,要做到一种未雨绸缪的效果,我们要先去除所有与婴儿有关的特征信息,仅仅是通过婴儿父母的基本信息以及婴儿的出生地来预测一下婴儿出生后存活的概率。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
selected_features = [
'INFANT_ALIVE_AT_REPORT' ,
'BIRTH_PLACE' ,
'MOTHER_AGE_YEARS' ,
'FATHER_COMBINED_AGE' ,
'CIG_BEFORE' ,
'CIG_1_TRI' ,
'CIG_2_TRI' ,
'CIG_3_TRI' ,
'MOTHER_HEIGHT_IN' ,
'MOTHER_PRE_WEIGHT' ,
'MOTHER_DELIVERY_WEIGHT' ,
'MOTHER_WEIGHT_GAIN' ,
'DIABETES_PRE' ,
'DIABETES_GEST' ,
'HYP_TENS_PRE' ,
'HYP_TENS_GEST' ,
'PREV_BIRTH_PRETERM'
]
births_trimmed = births . select ( selected_features )
在这个数据集中,大量的变量特征值都是Yes/No/Unknown值,我们将Yes编码成1,另外的其他值编码成0。
而在代表怀孕妈妈的吸烟数量的这个特征值的编码上,我们采用这样的规则。0:代表妈妈在怀孕期间没有抽过烟;而1-97:代表妈妈在怀孕期间真实的抽烟次数,而98:则代表孕期抽烟次数高达98次及以上;但99:意味着妈妈的孕期抽烟情况未知。
1
2
3
4
5
6
7
8
9
import pyspark.sql.functions as func
def recode ( col , key ):
return recode_dictionary [ key ][ col ]
def correct_cig ( feat ):
return func \
. when ( func . col ( feat ) != 99 ,
func . col ( feat )) \
. otherwise ( 0 )
rec_integer = func . udf ( recode , typ . IntegerType ())
由于Spark的机制问题,我们无法直接将DataFrame来用recode函数进行处理,所以我们首先要先它转换成Spark能够理解的UDF。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
births_transformed = births_trimmed \
. withColumn ( 'CIG_BEFORE' , correct_cig ( 'CIG_BEFORE' )) \
. withColumn ( 'CIG_1_TRI' , correct_cig ( 'CIG_1_TRI' )) \
. withColumn ( 'CIG_2_TRI' , correct_cig ( 'CIG_2_TRI' )) \
. withColumn ( 'CIG_3_TRI' , correct_cig ( 'CIG_3_TRI' ))
cols = [( col . name , col . dataType ) for col in births_trimmed . schema ]
YNU_cols = []
for i , s in enumerate ( cols ):
if s [ 1 ] == typ . StringType ():
dis = births . select ( s [ 0 ]) \
. distinct () \
. rdd \
. map ( lambda row : row [ 0 ]) \
. collect ()
if 'Y' in dis :
YNU_cols . append ( s [ 0 ])
最后,为了一次性转换所有的 YNU_cols 数据,我们用以下的方法:
exprs_YNU = [
rec_integer(x,
func.lit('YNU' )).alias(x)
if x in YNU_cols
else x
for x in births_transformed.columns
]
births_transformed = births_transformed.select(exprs_YNU)让我们来检查一下转换的结果吧:
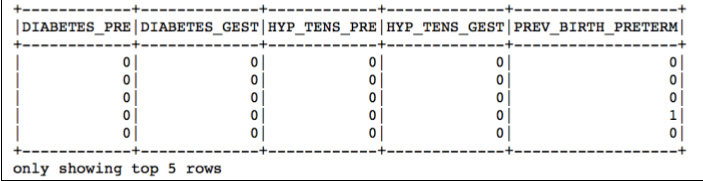
births_transformed.select(YNU_cols[-5:]).show(5)
数据预分析
为了建立一个良好的统计模型,我们首先需要了解清楚数据的组成分布以及背后的含义。
下面我们可以通过Spark提供的一些函数来对数据进行描述性分析。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
import pyspark.mllib.stat as st
import numpy as np
numeric_cols = [ 'MOTHER_AGE_YEARS' , 'FATHER_COMBINED_AGE' ,
'CIG_BEFORE' , 'CIG_1_TRI' , 'CIG_2_TRI' , 'CIG_3_TRI' ,
'MOTHER_HEIGHT_IN' , 'MOTHER_PRE_WEIGHT' ,
'MOTHER_DELIVERY_WEIGHT' , 'MOTHER_WEIGHT_GAIN'
]
numeric_rdd = births_transformed \
. select ( numeric_cols ) \
. rdd \
. map ( lambda row : [ e for e in row ])
mllib_stats = st . Statistics . colStats ( numeric_rdd )
for col , m , v in zip ( numeric_cols ,
mllib_stats . mean (),
mllib_stats . variance ()):
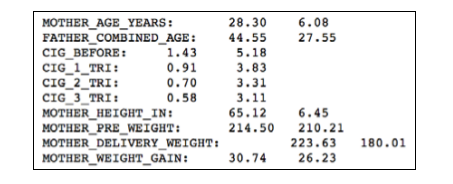
print ( '{0}: \t {1:.2f} \t {2:.2f}' . format ( col , m , np . sqrt ( v )))
根据输出的统计结果我们可以看出:在婴儿父母的年龄对比上,妈妈是明显比爸爸年轻的。妈妈的平均年龄在28岁左右,而爸爸的平均年龄确是44岁。
对于大部分的分类变量,我们也可以一一的来统计下他们的各个数值出现的频数:
1
2
3
4
5
6
7
8
9
10
11
12
13
categorical_cols = [ e for e in births_transformed . columns
if e not in numeric_cols ]
categorical_rdd = births_transformed \
. select ( categorical_cols ) \
. rdd \
. map ( lambda row : [ e for e in row ])
for i , col in enumerate ( categorical_cols ):
agg = categorical_rdd \
. groupBy ( lambda row : row [ i ]) \
. map ( lambda row : ( row [ 0 ], len ( row [ 1 ])))
print ( col , sorted ( agg . collect (),
key = lambda el : el [ 1 ],
reverse = True ))
根据这次的结果,我们又可以看出大部分的婴儿都是在医院出现的(医院的出生地代号BIRTH_PLACE=1)
相关系数
相关性的分析有利于我们发现特征变量中的多重共线性的情况,而多重共线性则是影响我们模型的鲁棒性的关键因素之一。
1
2
3
4
5
6
7
8
9
10
corrs = st . Statistics . corr ( numeric_rdd )
for i , el in enumerate ( corrs > 0.5 ):
correlated = [
( numeric_cols [ j ], corrs [ i ][ j ])
for j , e in enumerate ( el )
if e == 1.0 and j != i ]
if len ( correlated ) > 0 :
for e in correlated :
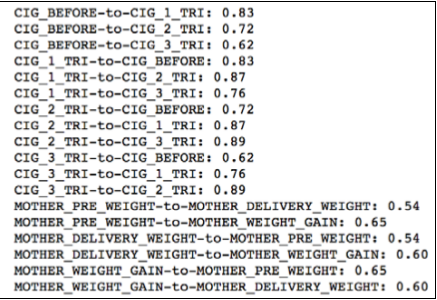
print ( '{0}-to-{1}: {2:.2f}' \
. format ( numeric_cols [ i ], e [ 0 ], e [ 1 ]))
上面的代码会替我们计算特征变量之间的相关系数矩阵,并输出相关系数高于0.5的特征。
根据上图输出的结果,我们又可以看出 CIG_XXX 系列的特征都有些非常高的相关性,所以在这个系列的特征之中保留一个即可。 在这里我只保留 CIG_1_TRI 这个特征。同理在WEIGHT系列中我只保留MOTHER_PRE_WEIGHT 这个特征。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
features_to_keep = [
'INFANT_ALIVE_AT_REPORT' ,
'BIRTH_PLACE' ,
'MOTHER_AGE_YEARS' ,
'FATHER_COMBINED_AGE' ,
'CIG_1_TRI' ,
'MOTHER_HEIGHT_IN' ,
'MOTHER_PRE_WEIGHT' ,
'DIABETES_PRE' ,
'DIABETES_GEST' ,
'HYP_TENS_PRE' ,
'HYP_TENS_GEST' ,
'PREV_BIRTH_PRETERM'
]
births_transformed = births_transformed . select ([ e for e in features_
to_keep ])
小结
在这一篇实战的文章中,我讲解了:
数据的载入和转换
数据的描述性分析
数据相关性分析
限于时间和篇幅,我打算将有关分类变量的统计检验分析,以及最后的特征选取和建模放在下一篇文章之中,欢迎大家继续阅读我的下一篇文件。